LINGÜÍSTICA DE INTERAÇÕESMOLECULARES[1]
Romeu Cardoso GUIMARÃES[2]
RESUMO: As moléculas biológicas mais interessantes são longos polúneros. Em analogia com alinguagemhumanaalfabltica,estes podem ser chamados de textos, e analisados, quanto à estrutura primária, como seqüência de letras (monômeros; como nucleotfdeos, aminoácidos, etc.) ou de palavras (códigos de oligômeros, de atl 5-ó letras).Considera-se que o estudo das palavras, em abordagem de tipo lingüfstico, possa contribuir para o entendimento das intérações (comunicações) moleculares. As linguagens e dialetos, moleculares e humanos, são contrastados� Alinguagem molecular se distingue peculiarmente da humana, por exemplo, por utilizar forma tridimensional, dinlimica temporal, ausência de espaçamento e pontuação, e sobreposição de significados. Apresenta-se um rnl todo matemático para descoberta. depalavrasemtextos.Apalavra AA (trinca de adeninas) foi estudada na evolução do RNA ribossômico 5S. Observou-se que esta palavra l mais freqüente em organismos menos complexos e menos freqüente nos mais complexos, das linhagens de fungos, plantas e vertebrados. Nas duas últimas, reduziu-se tamblm o grau de variabilidade gênica. Pelo contrário, grau moderado de freqüência da palavra persistiu em toda a linhagem dos invertebrados, com manutenção paralela de alto nlvel de variabilidade gênica. Nas mitocôndrias, plastfdeos e micoplasmas, a freqüência da palavra AAA foi aumentada, de acordo com sua necessidade de interações com maior amplitude de variação. Esses comportamentos indicam que a palavra monótona AA pennite ambigüidade de interações. Com a evolução da complexidade orgânica e da maior especificidade molecular, as palavras amb(guas foram progressivamente evitadas.
UNITERMOS: Bioqu(mica; polúneros; palavras; códigos; interações; lingü(stica; comunicação.
Apresento alguns procedimentos teóricos sobre a análise de seqüências de polímeros biológicos que interessam ao tema das interações moleculares.
Esses trabalhos se baseiam em princípios análogos aos de alguns estudos da linguagem humana. A analogia não é extensa, porque a "linguagem molecular" apresenta muitas peculiaridades e distinções. No entanto, o estabelecimento de contrastes com algo que nos é familiar (neste caso, a linguagem ocidental, baseada no alfabeto) pode auxiliar na compreensão do problema.
Um exemplo de interações moleculares é ilustrado (Fig. 1) com o caso do Citocromo C. Note-se que a molécula possui uma conformação espacial, ou tridimensional, que demarca um centro ativo, interno, com a propriedade (função) de coordenar a ligação de uma porfIrina (heme). Esta, por sua vez, coordena a ligação de um metal, que é o responsável imediato pela função.
A molécula possui, ainda, propriedades de interação com outras moléculas, no caso, por exemplo, para ancoragem à membrana mitocondrial, através de regiões externas da molécula.
Esses princípios são comuns aos vários outros casos de interações moleculares, como: enzima-substrato, antígeno-anticorpo, indutor-molécula alvo, hormônio-receptor, e os casos de agonistas, antagonistas e medicamentos com seus receptores , etc.
A abordagem tradicional, ilustrada na Figura 1, privilegia o estudo de letras, ou monômeros , como nos estudos de mutações puntiformes, por troca de aminoácidos singulares. A abordagem lingüística apresenta, como principal novidade, o estudo de palavras, em vezdeletras.A analogia com a linguagemhumanaalfabéticapropõe a terminologia de sentenças ou textos para as moléculas inteiras, de palavras para pequenos segmentos das moléculas (oligômeros, tipicamente com cerca de 5-ó elementos; (15)), e de letras para os elementos síngulares (aminoácidos, nas proteínas ; nucleotídeos , nos ácidos nucléicos, RNA ou DNA; e assim por diante) .
ANALOGIAS ENTRE AS LINGUAGENS HUMANA E MOLECULAR
Passo, então, a ressaltar os detalhes que fazem se assemelhar ou distinguir as linguagens humana e molecular.
De início, a molecular é espacial e conformacional (Fig. 1), enquanto a humana é linear. A configuração global do sentido de uma sentença humana só é apreendida totalmente, com forma "gestáltica", ao flm da leitura seqüencial.
A humana tem pontuaçãoe espaçamento entre palavras e sentenças. Por exemplo, o espaçamento adequado, segundo o idioma inglês, é que nos permite entender o sentido da seqüência de letras justapostas togethernowhere (Fig. 2), que pode ser lida de 4 maneiras distintas. Na molecular a justaposição é a regra e seu desdobramento em palavras é difícil.
Ambas são degeneradas, com sinonímia freqüente, como é o caso dos codons para tradução de mRNA em proteínas (l O).
A linguagem molecular é altamente superposta; o mesmo texto pode ser lido de várias maneiras não-sinônimas, com elevada densidade ou compactação da informação (Fig. 3). O mesmo segmento de DNA codillca várias funções, cada uma de suas palavras pode significar informações diferentes. O DNA codiflca interações com proteínas, para regulação gênica e constituição da cromatina. Está, também, embutida no DNA, a informação necessária para que o RNA, transcrito dele, desenvolva sua estruturação secundária e terciária e interaja com proteínas , para processamento e regulações.
o DNA pode, ainda, produzir diferentes RNAs, dependendo do modo como é transcrito e processado. Até um único RNA pode ser traduzido de modos alternativos, produzindo proteínas diferentes (1 1). A alta ambigüidade que se detecta ao nível dos genes é somente reduzida nas proteínas. Mesmo as enzimas apresentam um certo grau de inespecificidade nas suas funções (ó).
A leitura da codificação molecular é, também, dinâmica e multidimensional, modulada por movimentos e freqüências, até com casos de ritmicidade que se assemelhariam à métrica da poesia e da música. Em todas as interações são importantes as variações de equilíbrios térmicos, expressas pela química como constantes de afinidade ou de associação e dissociação, cujas alterações podem afetar grandemente os resultados (12). Por exemplo, (Fig. 4) há uma periodicidade modular estatística NNG nos rnRNA, que corresponde a uma periodicidade complementar NNC em sítios do rRNA; há, também, uma periodicidade estatística de AA no DNA, a cada 10,5 bases, que marca curvaturas em passos regulares da dupla hélice e interações do DNA com proteínas cromatínicas . Essas periodicidades são residuais, ocultas sob as especificidades de cada seqüência.
![]() O nosso alfabeto tem mais de
20 letras, mas o som de cada uma pode variar, conforme o contexto silábico em
que se situa, gerando mais de 40 sons (Quadro 5). Semelhantemente, a codificação molecular usa 4 letras fundamentais, as 4 bases primárias dos
ácidos nucléicos, chegando a pouco mais de 20 aminoácidos, nas proteínas , a reatividade de cada elemento diferirá,
conforme suas vizinhas na "palavra" molecular. Estas são chamadas de
palavras-códigos e seu sentido pode variar conforme
o ambiente térmico,iônico e hidropático. Com a mudança em uma palavra, às vezes toda a sentença (polúnero)sealtera; por
exemplo, no caso das proteínas com
propriedades alostéricas (9), ou
em algumas trocas de aminoácidos, como na Hemoglobina
S.
Com a troca de um único
aminoácido, em ambiente pobre em oxigênio,
a hemoglobina adquire
solubilidade e conformação alterada, que produz a hemácia
falciforme (1 ó) .
O nosso alfabeto tem mais de
20 letras, mas o som de cada uma pode variar, conforme o contexto silábico em
que se situa, gerando mais de 40 sons (Quadro 5). Semelhantemente, a codificação molecular usa 4 letras fundamentais, as 4 bases primárias dos
ácidos nucléicos, chegando a pouco mais de 20 aminoácidos, nas proteínas , a reatividade de cada elemento diferirá,
conforme suas vizinhas na "palavra" molecular. Estas são chamadas de
palavras-códigos e seu sentido pode variar conforme
o ambiente térmico,iônico e hidropático. Com a mudança em uma palavra, às vezes toda a sentença (polúnero)sealtera; por
exemplo, no caso das proteínas com
propriedades alostéricas (9), ou
em algumas trocas de aminoácidos, como na Hemoglobina
S.
Com a troca de um único
aminoácido, em ambiente pobre em oxigênio,
a hemoglobina adquire
solubilidade e conformação alterada, que produz a hemácia
falciforme (1 ó) .
![]() Os mais longos segmentos
dos ácidos nucléicos que interagem com proteínas chegam a 20-30
bases (15), como algumas palavras hu
as. Também, em ambas situações, as palavras mais comuns têm cerca
de 5-ó letras (Quadro
ó). Os vocabulários são pequena fração do total de combinações possíveis, portanto,
com alto grau de seletividade e, como corolário, de repetitividade. Esta última, em eucariotos complexos, varia de 1 a milhões
(8).
Os mais longos segmentos
dos ácidos nucléicos que interagem com proteínas chegam a 20-30
bases (15), como algumas palavras hu
as. Também, em ambas situações, as palavras mais comuns têm cerca
de 5-ó letras (Quadro
ó). Os vocabulários são pequena fração do total de combinações possíveis, portanto,
com alto grau de seletividade e, como corolário, de repetitividade. Esta última, em eucariotos complexos, varia de 1 a milhões
(8).
O trabalho de decifrar a codificação molecular é difícil, com procedimentos laboratoriais e computacionais demorados e tediosos. O montante atualmente conhecido não chega a 1 % do tamanho total do genoma de mamíferos (7). A maior parte desse total conhecido é composto pelos ó4 tipos de trincas que constituem os codons para tradução de proteínas.
A LINGUAGEM GeNÔMlCA
A linguagem genética foi chamada por Trifonov e Brendel (15) de GNÔMICA, derivada do grego (gnomon: máxima, norma, aforisma) , também usada para denominar os ponteiros dos relógios solares e os gnomos ou duendes da mitologia nórdica (semelhantes aos homúnculos que os primeiros microscopistas diziam enxergar nos espermatozóides?), e que se adequa bem aos genes, como em genoma e em "genomês" . O dicionário gnômico de 198ó continha cerca de 800 palavras.
Algumas palavras gnômicas são praticamente universais, consensuais para muitos tipos de organismos (Fig. 7).
![]() Outros vocabulários compõem
grupos lingüísticos (dialetos) menores como: os sítios de restrição que são
usados na engenharia genética, funcionam
in vivo nas bactérias (4) ; os genes dos rRNA e das rPROT (proteínas
ribossômicas) têm vocabulários
semelhantes, indicando convergência, por compartilharem funções
e regulações, ou homologia ancestral;
bactérias e seus bacteriófagos simples também usam os mesmos dialetos,
novamente indicando convergência, por adaptação parasitohospedeiro, ou origem comum;
etc. (13).
Outros vocabulários compõem
grupos lingüísticos (dialetos) menores como: os sítios de restrição que são
usados na engenharia genética, funcionam
in vivo nas bactérias (4) ; os genes dos rRNA e das rPROT (proteínas
ribossômicas) têm vocabulários
semelhantes, indicando convergência, por compartilharem funções
e regulações, ou homologia ancestral;
bactérias e seus bacteriófagos simples também usam os mesmos dialetos,
novamente indicando convergência, por adaptação parasitohospedeiro, ou origem comum;
etc. (13).
PALAVRAS-CONTRASTES
Um método matemático foi desenvolvido pelo grupo de Trifonov para descobrir palavras-códigos em textos genéticos, que estamos agora começando a aplicar a estudos do rRNA 5S. O princípio do método é o das cadeias de Markov , simples e de apreensão intuitiva (Fig. 8).
O método não é estritamente estatístico ou probabilístico porque não depende fundamentalmente da freqüência de ocorrência das palavras. Tem,ainda,as vantagens de independer de modelos paramétricos e dos tamanhos ou de homologias entre as moléculas a serem comparadas .
Calcula-se o valor do contraste (proporção) entre a freqüência observada de uma palavra no texto com a freqüência esperada, a partir de seu segmento interno.Por isso, as palavras identificadas pelo método são chamadas de palavras-contrastes.
Um contraste elevado (até 1) significa correlação alta entre as letras anterior e posterior com o segmento interno ; sugere que a palavra é confiável, tem boas chances de ser demonstrada como palavra real, e merece ser investigada experimentalmente. No caso exemplificado, toda vez que se colocar R após ELHO, será obrigatório colocar-se M antes, e MELHOR será boa palavra-contraste.
Constroem-se vocabulários de palavras-contrastes. A investigação experimental confirmará a validade das palavras e, quando sua função e significado forem descobertos, poder-se-á construir os dicionários semânticos.
A PALAVRA AAA NO rRNA 5S
Passo, agora, a apresentar resultados de nosso trabalho(3)sobre a palavra AAA no rRNA 5S. Este RNA é pequeno, com somente 120 bases, mas apresenta a grande vantagem de ser ubiquitário; seu texto já foi decifrado em cerca de ó00 moléculas, ao longo de toda a evolução.
O trabalho é somente evolutivo e comparativo. Portanto, não há dados sobre o significado semântico da palavra. No entanto, conseguimos desenvolver uma interpretação sobre algumas de suas propriedades informacionais.
As freqüências de ocorrências da palavra, nas principais linhagens de organismos (Fig. 9), produziram regularidades que foram interpretáveis porcomparaçãocom outros dados biológicos.
![]() O
modelo interpretativo utilizado é
de que a palavra AAA possui ambigüidade informacional na interação com outras moléculas , como as proteínas (Fig. 1 0). Diz-se que, quando um aminoácido de uma proteína deve interagir
especificamente com uma adenina do
RNA, e esta adenina é ladeada por outras bases diferentes, a palavra (trinca) é
complexa e a interação será posicionalmente inambígua, específica ou unívoca. Por outro lado, quando a adenina é ladeada
por outras adeninas , a interação peecerá qualitativamente específica, mas se
tomará posicionalmente ambígua. O RNA tolerará a interação com a I !!, 2!! ou 3!! adenina igualmente, possibilitando deslocamentos e aceitando
variações na posição
dos aminoácidos, de até 3 para a frente ou para trás .
O
modelo interpretativo utilizado é
de que a palavra AAA possui ambigüidade informacional na interação com outras moléculas , como as proteínas (Fig. 1 0). Diz-se que, quando um aminoácido de uma proteína deve interagir
especificamente com uma adenina do
RNA, e esta adenina é ladeada por outras bases diferentes, a palavra (trinca) é
complexa e a interação será posicionalmente inambígua, específica ou unívoca. Por outro lado, quando a adenina é ladeada
por outras adeninas , a interação peecerá qualitativamente específica, mas se
tomará posicionalmente ambígua. O RNA tolerará a interação com a I !!, 2!! ou 3!! adenina igualmente, possibilitando deslocamentos e aceitando
variações na posição
dos aminoácidos, de até 3 para a frente ou para trás .
Este modelo nos permitiu oferecer uma interpretação para a variabilidade (polimorfismo ou heterozigosidade de alelos) gênica observada nos invertebrados, que é o dobro (47% de genes polimórficos) da apresentada pelos vertebrados e plantas (25-2ó% ; (1» . Nossos dados sugerem que o mesmo, das duas últimas rotas, deve ter ocorrido na evolução dos fungos complexos . Nessas rotas evolutivas, as palavras ambíguas foram selecionadas contra, foram reduzidas ou evitadas. O RNA deve ter desenvolvido outras interações ao lado ou superpostas às das palavras ambíguas , tornou-se mais caregado de funções, e a permanência das palavras ambíguas foi prejudicial às novas interações. As palavras ambíguas foram substituídas por outras mais complexas . Os invertebrados não seguiram essas rotas , mantiveram as palavras ambíguas e a tolerância a maior grau de polimorfismo gênico.
O inverso ocorreu na evolução das organelas (mitocôndrias e plastídeos) e dos micoplasmas, que aumentaram a freqüência de utilização das palavras ambíguas. Pelo menos para ocaso das organelas , a explicação é consistente com os dados biológicos e a teoria de sua origem endossimbiôntica (14). Atualmente, as rPROT que interagem com seus rRNA são, na grande maioria, nucleares. No entanto, deve ter havido um período de adaptação das bactérias à associação com a célula eucariótica, quando o RNA da bactéria de vida livre foi forçado a interagir com as rPROT nucleares. Estas são homó10gas às bacterianas, as diferenças entre os dois tipos são maiores que entre variantes alélicas, e o acúmulo das palavras ambíguas foi selecionado a favor.
Existe, ainda, para essa rota de aumento da freqüência das palavras ambíguas, a possibilidade de evolução neutra (5), por relaxamento de pressões seletivas. ORNA da bactéria associada ao eucarioto pode, simplesmente, ter perdido interações com proteínas, possibilitando acúmulo aleatório de adeninas ao longo de todo seu genoma. Nossos dados, especialmente sobre as mitocôndrias (3), não favorecem essa possibilidade, mas o caso dos micoplasmas poderia ser consistente com a hipótese da evolução neutra. Essas bactérias são extracelulares, aderidas às membranas plasmáticas (2), e poderiam não utilizar rPROT nucleares para compor seus ribossomos. Estamos à procura de dados experimentais para decidir entre essas duas possibilidades.
Em conclusão, as principais indicações desse estudo são:
1. o aumento da complexidade lingüística (ou a redução da ambigüidade informacional) molecular foi paralelo à redução de tolerância à variabilidade gênica;
2. nas linhagens de vertebrados e plantas (e fungos) complexos houve aumento paralelo da complexidade molecular;
3. na linhagem dos invertebrados, manteve-se nível moderado de complexidade molecular;
4. em organelas endossimbiônticas e micoplasmas (parasitas celulares obrigatórios), amplificou-se a ambigüidade informacional.
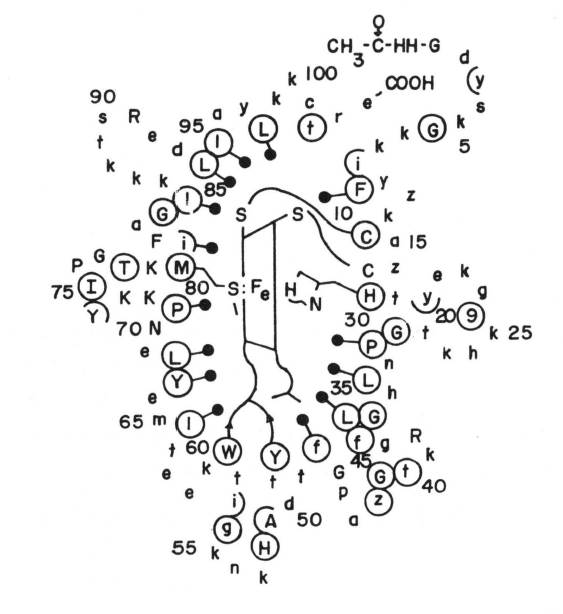
Figura 1. Acondicionamento do anel porfrrínico (heme) na·molécula do Citocromo C. Os resíduos de aminoácidos com círculos têm suas cadeias laterais voltadas para o interior da molécula. As "cabeças de alrmetes" assinalam os resíduos em contato com o heme. Os semicírculos indicam os resíduos com cadeias laterais parcialmente voltadas para o interior da molécula. As setas partindo da tirosina 48 e do triptofano 59 representam pontes de hidrogênio. Os resíduos em maiúsculas são os que permaneceram invariáveis em 29 espécies. Os aminoácidos estão indicados pelo código de letras singulares, em vez de trincas. O átomo de ferro, no centro do heme, é coordenado pela 'cadeia lateral da histidina 18 e pelo enxofre da metionina 80. Note-se que 24 dos 39 resíduos marcados com círculos ou semicírculos aparecem em "palavras" de 2 ou 3 "letras". Adaptado de: R. Acher -1974- "L'évolution moléculaire au niveau des protéines!" Biochimie, v. 5ó, p. 1-19.
"togethemowhere "
together nowhere
together now here
to get her nowhere
to get her now here
Figura 2. Espaçamento entre palavras na linguagem humana alfabética. O exemplo apresentado é do idioma inglês. A seqüência de 15 letras justapostas pode ser lida de até 4 maneiras distintas, conforme os espaçamentos utilizados. Adaptado de: E.N. Trifonov -1989- "The multiple codes of nucleotide sequences." Buli. Math. Biol., v. 51, p. 417-32.
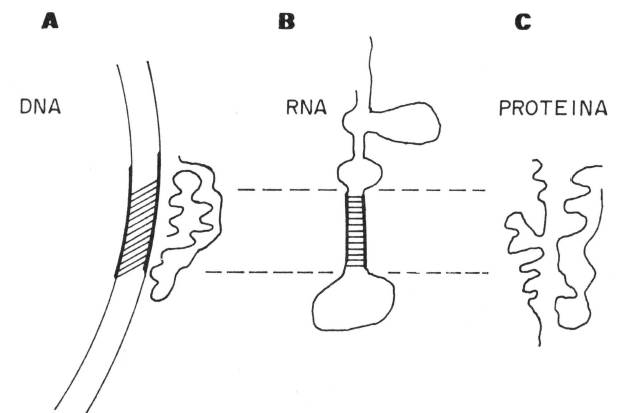
Figura 3. Superposição de mensagens na linguagem molecular. Um segmento de DNA contém até 3 níveis (classes, tipos) de mensagens sobrepostos: (a) para interação do DNA com proteÚlas na estruturação da cromatina e nas regulações de transcrição; (b) codificando a estrutura secundária e terciária do RNA transcrito, e a interação deste com proteínas na regulação e no processamento; (c) codificando a estrutura secundária e terciária das proteínas traduzidas, suas associações quaternárias, modificações pós-traducionais e funções. Adaptado de: E.N. Trifonov -1988-'Codes of nuc1eotide sequences." In: Non linearity in biology and medicine. Eds: A.S. Perelson, B. Goldstein, M. Dembo and l.A. laques. Elsevier, New York. Mathematical Biosc., v. 90, p. 507-17.
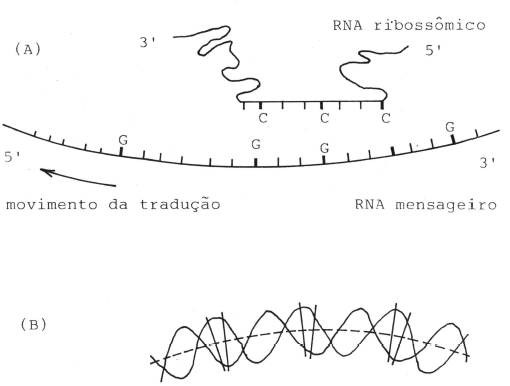
Figura 4. Códigos ocultos e periódicos nas seqüências nuc1eotídicas. Esses códigos são genéricos e estatísticos, descobertos após análise de periodicidades remanescentes na estrutura de grande número de seqüências, heterogêneas quanto às funções primárias e específicas. (a) As seqüências dos RNA mensageiros têm, mais freqüentemente, Guaninas nas primeiras posições das trincas codônicas. Alguns sítios do RNA ribossômÍco têm Citosinas espaçadas precisamente em posições Cn, Cn + 3 e Cn +ó, e localizados em regiões do ribossomo que interagem com os RNA mensageiros, que funcionam como marcadores dos módulos das trincas, no processo da tradução. (b) A dupla hélice do DNA apresenta uma curvatura intrínseca que depende da presença, mais freqüente, das bases vizinhas AA (ou TI), em intervalos regulares de 10,5 bases. Este intervalo corresponde a um passo completo da dupla hélice. A curvatura intrínseca facilita o enrolamento do DNA em tomo das proteínas que compõem os nuc1eossomos da cromatina. Os sítios das duplas AA (ou TI) são os que presentam curvatura mais acentuada. AdaptadodeE.N.Trifonov -1989- "The multiple codes of nuc1eotide sequences." Buli. Math. Biol. , v.51, p. 417-32.
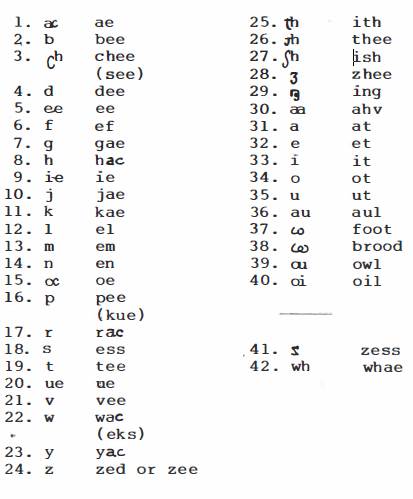
Quadro 5. As letras do alfabeto e os códigos de sons.
O exemplo é do idioma inglês. Diferentes combinações de letras produzem contextos silábicos distintos nos quais a mesma letra participa de sons diferentes. Assim, o "alfabeto" sonoro é mais extenso que o conjunto das letras individuais. Adaptado de: D. Diringer -1 9ó8- The alphabet. A key tothe
history ofmankind.3.ed., 2 vol., Hutchinson, London, p.424.
![]() Quadro
6. Repetitividade e especificidade na linguagem humana.
Quadro
6. Repetitividade e especificidade na linguagem humana.
As obras completas de Shakespeare, segundo análise de B. Efrom e R. Thisted (1976 "Estimatingthenumberof unseenspecies: howmanywordsdid Shakespeare know? "Biometrikav.63,p.435-57),contêmvocabuláriode 31 .534 palavras. Para o total de 884.ó47 palavras escritas, obteve-se tamanho médio de 5 letras por palavra e repetitividade média de 28 ( btal de palavras/vocabulário) . A distribuição aleatória das 2óletras, em grupos de 5, produz 12 x 10ó palavras (seletividade de 14x), com repetitividade de 1 ,07.
As 4 letras dos ácidos nucléicos produzem 1ó palavras de 2 letras , ó4 de
3 letras (como os codons), 25ó de 4 letras, 1.024 de 5 letras e 4.096 palavras de
6 letras. Ver: E.N. Trifonov -1988- "Nucleotide sequences as a language : morphological classes of words." In: Classification and related methods 01 data analysis.Ed.H.H. Bock, Elsevier, North Holland, p. 57-64.
![]() Figura 7. Alguns exemplos
de palavras GeNÔMICAS consensuais.
Figura 7. Alguns exemplos
de palavras GeNÔMICAS consensuais.
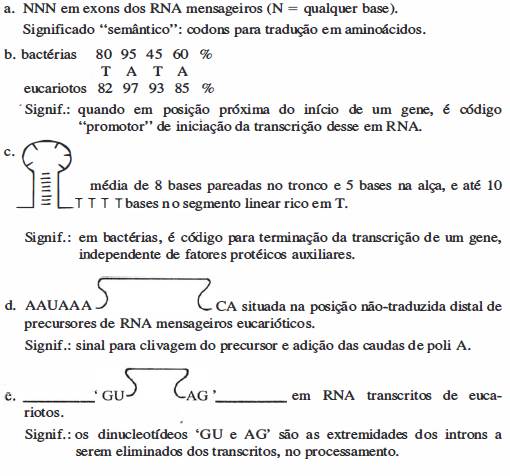

Compilado de várias fontes: ver R.C. Guimarães -1987- Estrutura e função do RNA.In:Genética molecular e de microorganismos. Ed.SOP Costa.Manole, São Paulo,p.39-77 ;BLewin -1 990-GenesIV.OxfordUniv.Press, Oxford UK, 857 p.; J.D. Watson, N.H. Hopkins, J.W. Roberts , J.A. Steitz & A.M. Weiner-4th ed. , Benjamin/Cummings, Menlo Park, Cal USA, 1987 1. 163, p.
Figura8. Ilustração do método do contrasteparaidentificação de palavras-códigos em polímeros.
O polúnero é considerado como um texto contínuo. Segmentos internos de tamanho 1 ou mais são a base para o teste das letras vizinhas, anterior e posterior. O exemplo é para uma palavra de ó letras , com segmento interno de 4 letras. As letras representadas por . são mais variáveis que as apresentadas .

O método foi desenvolvido por V. Brendel, J.S. Beckmann e E.N. Trifonov -198ó- Linguistics of nucleotide sequences: morphology and comparison of vocabularies. J. Biamo/ec. Struct. Dynam ." v. 4, p. 11 -21.
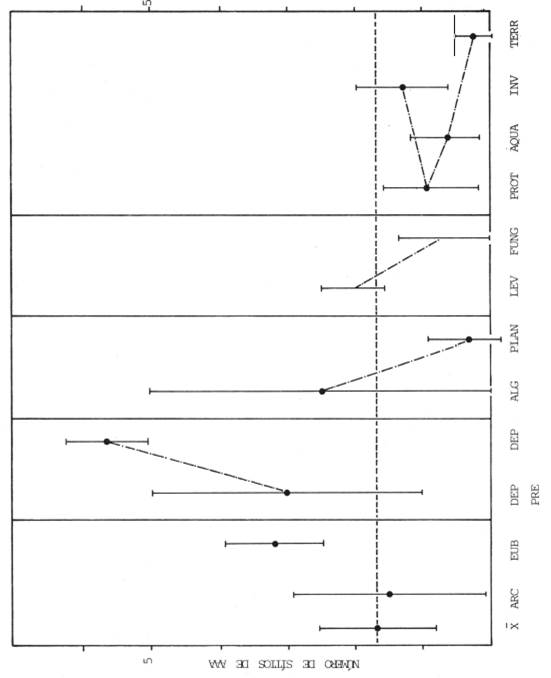
Figura 9. Freqüência das palavras AAA no RNA ribossômico 5S, ao longo da evolução.
São apresentados os números inédios de ocorrência de sítios de agrupamentos de adeninas (trincas ou mais longos), por grupo de organismos, dentro das categorias apresentadas.
![]() X =média geral de ocorrência de AA por grupo, em todas as categorias ; ARC, arquebactérias; EUB , eubactérias ; DEP PRE, eubactérias de vida liprecursoras dasDEP;DEP, organelas de eucariotos (mitocôndrias e
plastídeos) e micoplasmas; ALGas; PLANtas
; LEVeduras e FUNgos das categorias
Ascomicetos e Basidiomicetos; PROTistas ; INVertebrados; grupos
AQUÁticos (incluindo anfíbios) e TERRestres da linhagem dos vertebrados. Os dados são médias e desvios padrão por categoria;
para LEV e FUNG, os dados são os limites
apresentados pelos grupos componentes das categorias. Dados extraídos de R.c. Guimarães e V.A. Erdman (1 990) .
X =média geral de ocorrência de AA por grupo, em todas as categorias ; ARC, arquebactérias; EUB , eubactérias ; DEP PRE, eubactérias de vida liprecursoras dasDEP;DEP, organelas de eucariotos (mitocôndrias e
plastídeos) e micoplasmas; ALGas; PLANtas
; LEVeduras e FUNgos das categorias
Ascomicetos e Basidiomicetos; PROTistas ; INVertebrados; grupos
AQUÁticos (incluindo anfíbios) e TERRestres da linhagem dos vertebrados. Os dados são médias e desvios padrão por categoria;
para LEV e FUNG, os dados são os limites
apresentados pelos grupos componentes das categorias. Dados extraídos de R.c. Guimarães e V.A. Erdman (1 990) .

Figura 10. Modelo explicativo da ambigüidade informacional dapalavra AAA no RNA ribossômico 5S .
À esquerda, uma adenina (A) é ladeada por outras bases (N) e a trinca é uma palavra complexa. Quando um anlÍnoácido (aa) de uma proteína deve interagir com a adenina, a interação será específica e posiciona1mente inambígua.
À direita, adeninas vizinhas
compõem uma palavra (trinca) monótona.
As inte![]() rações dos anlÍnoácidos
podem pe ecer específicas com as adeninas,mas tomam-se posicionalmente ambíguas. A trinca tolerará interações com
proteínas, onde o anlÍnoácido pode estar deslocado em até, 3 posições, por
inserções ou deleções na seqüência. Extraído de R.C. Guimarães e V.A. Erdman (1 990) .
rações dos anlÍnoácidos
podem pe ecer específicas com as adeninas,mas tomam-se posicionalmente ambíguas. A trinca tolerará interações com
proteínas, onde o anlÍnoácido pode estar deslocado em até, 3 posições, por
inserções ou deleções na seqüência. Extraído de R.C. Guimarães e V.A. Erdman (1 990) .
AGRADECIMENTOS
CNPQ,FUNDUNESP E SocoAmigos Inst. Weizmann em São Paulo.
GUIMARÃES,R.C.Linguisticsofmolecularinteractions. Trans/Form/Ação, São Paulo, v. 14, p. 123-137, 1991.
ABSTRACT: The most interesting biological molecules are long polymers. In analogy with human alphabetic languages,they can be called texts and analysed, as to the primary structure, as sequences of letters (monomers; nucleotides, aminoacids, etc.) or of words (codes of oligomers, of up to 5- ó letters) . It is considered that the study of words, in a linguistic approach, may contribute positively to the understanding of molecular interactions (communication). The molecular and human languages and dialects are contrasted. The molecular one is peculiarly distinct fromthehuman, for instance,by itsuse of a tridimensional morphology,temporal dynamics, absence of spacings and punctuation, and overlapping messages. A mathematical method is presented, for discovering words in texts. The word AA (adenine triplets) was studied in the evolution of the 5S ribosomal RNA. It was shown that this word is more frequent in less complex organisms and less frequent in the more complex ones, in the fungi, plants, and vertebrates lineages. In the two latter ones, the degree of genic variability was also reduced. To the contrary, a moderate degree of usage of this word persisted in the whole invertebrates lineage, where a high degree of genic variability was maintained. In mitochondria, plastids and mycoplasmas, the frequency of the word AA was increased, consistently with their need for interactions with a wider range of variation. These behaviors indicate that the monotonous AA word allows for ambiguity in interactions. With the evolution of organic complexity and of greater molecular specificity, ambiguous words were progressively avoided.
KEYWORDS: Biochemistry; polymers; words; codes; interactions; linguistics; communication.
REFERÊNCIASBmLIOGRÁFICAS
1. AYALA, F. J.,KIGER Jr., J. A.Modem genetics.MenloPark:BenjaminlCuInIngs, 1980. p.ó22.
2. GHOSH, A., DAS 1., MANILOFF, J. Lack of repair of ultraviolet light damage in Mycopúisma gallisepticum. Journal of molecular Biology, London, v. 11 ó, p. 337- 344, 1977.
3. GUIMARÃES, R. C., ERDMANN, V. A.Evolution adenineclustering in 5S rRNA.1990. (Texto mimeografado).
4. KESSLER, C., NEUMAIER, P. S., WOLF, W. Recognition sequences of restriction endonucleases and methylases: a review. Gene, Amsterdan, v. 33, p. 1- 102, 1985.
5. KIMURA, M. The neutral theory of molecular evolution. Cambridge: Cambridge University Press, 198ó.
6. KIRKWOOD, T. B. L., ROSENBERGER, R.F., GALAS,D.1., ed. Accuracyin molecular processes: its control and relevance to living systems. London: Chapman & Hall, 198ó. 391p.
7. MCKUSICK,V.A.Mendelian inheritance in mano 7ed.B altimore:TheJohnsHopkins University Press, 198ó. p. xvü-xviii.
8. MIKLOS, G. L. G. Localized highIy repetitive DNA sequences invertebrateand invertebrate genomes. In: MACINTIRE, R. J., ed.- Molecular evolutionary genetics. New York: Plenum Press, 1985. p. 24 1-321.
9. MONOD, J., CHANGEUX, J. P., JACOB , F. Allosteric proteinsandcellularcontrol systems. Journal ofmolecular Biology, London, v. ó, p. 30ó-329, 19ó3.
10. NIRENBERG, M. W., JONES, O.W.,LEDER,P., eta lo On the coding of genetic information. In:ColdSpringHarborSymposium onQuantitativeBiology,28, p. 549-557. 1 9ó3.
11. PARDINI, M.I.M.C., GUIMARÃES,R. C.Um conceito sistêmico-funcional do gene. Botucatu: Instituto de Biociências da UNESP,1989. Dissertação (Mestrado).
12. PERELSON, A. S., OSTER, G. F. Theoretical studies of Donal selection: minimal antibody repertoire size and reliability of self-non- self discrimination. Journal of theoretical Biology, v. 81, p. ó45-70. 1979.
13. PIETROKOVSKI, S., HIRSHON, J., TRIFONOV, E.N.Linguisticmeasureof taxonomic and functional relatedness of nucleotide sequences. 1990. (Texto mimeografado) .
14. RAZIN, S., FREUNDT E. A. The mycoplasmas. In: Bergey's manual of systematic bacteriology I. B altimore: Williams and Wilkins, Krieg, N.R., ed., p. 740-793. 1984.
15. TRIFONOV, E. N., BRENDEL, V. Gnomic: adictionary of genetic codes. B alaban: Rehovot, 31 7p.
16. WEATHERALL, D. J., CLEGG, J. B., HIGGS . D.R., WOOD,W.G.The hemoglobinopathies. In: SCRIVER,C.R., BEAUDET, A.L., SLY,W. S., VALLE, D., ed. Themetabolic basis ofinherited disease.New York: McGraw-Hil, 1989. p. 2 281- 2 339.